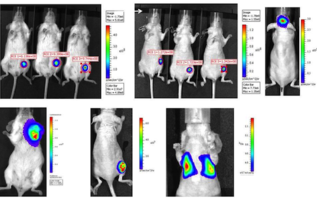
Imagerie in vivo
Principe
L'imagerie in vivo est la visualisation non invasive d'organismes vivants à des fins de recherche ou de diagnostic. On y retrouve en général deux domaines clés : imagerie anatomique/morphologique et imagerie moléculaire. En imagerie moléculaire, les fonctions cellulaires ou les procédés moléculaires sont visualisés, généralement à l'aide de biomarqueurs. En imagerie anatomique, aucun marqueur n'est utilisé et la visualisation est basée sur les propriétés intrinsèques des tissus et des organes à observer, comme l'atténuation des rayons X lors de la tomographie assistée par ordinateur.
Avec l'imagerie moléculaire in vivo, plusieurs événements peuvent être suivis.
- Suivi d’évènements biologiques (moléculaires, cellulaires, métaboliques, génétiques, …)
- Analyses structurales et/ou fonctionnelles
- Suivi de processus physiopathologiques (croissance tumorale, inflammation,
infection, …) - Suivi de cellules / agent pathogène, interactions
- Biodistribution de molécules
- Evaluation de l’efficacité de candidats thérapeutiques
- Etudes pharmacodynamiques, pharmacocinétiques
- …
Par rapport aux autres méthodes de mesure des procédés moléculaires, l'imagerie in vivo présente plusieurs avantages majeurs:
- Elle fournit la localisation spatiale du procédé ou de la molécule.
- Elle est dynamique: les changements au fil du temps peuvent être suivis.
- Elle peut être répétée plusieurs fois chez un même individu.
- Elle permet d'observer un procédé (principalement) non perturbé, c'est-à-dire que les résultats sont physiologiquement pertinents.
Ces avantages font de l'imagerie in vivo une technologie très utile dans de nombreux domaines de recherche et de diagnostic.
Instruments disponibles :
Campus Pasteur
Description :
- Marque : IVIS Lumina XRay (Perkin Helmer)
- Logiciel : Living Image 4.3.1
- Excitations : lampe de fluorescence SHOPT (Visible et IR, privilégier les longueurs d’ondes IR)
- Emissions : standard Emission Filter wheel et Low Range Emission filter wheel (500 à 620nm)
- Modalité : Photographie | Bioluminescence | Fluorescence | Radiographie/RayonsX
- TL : BF
- Détecteurs : CCD Andor (3 souris / prise de vues)
- Chambre d'incubation Temp. et CO2 : Okolab
- Environnement : platine chauffé, système d’anesthésie gazeux à l’isoflurane
- Accessoires : acquisition multi spectrale en fluorescence
- A2 : communication par passe plat avec le A2 et maintien du confinement en boite Biospace type 3
Applications :
- Visualisation d’évènements biologique sur petit animal corps entier, de manière non invasive, au cour du temps. En adéquation avec les 3Rs :
- Études longitudinales
- Statistiques : moins de variations
- « Réduction » du nombre d’animaux nécessaires
- Méthodes non-invasives
- « Raffinement »
-
Applications en recherche préclinique :
- Suivi de processus physiopathologiques (croissance tumorale, inflammation,
infection, …) - Suivi d’évènements biologiques (moléculaires, cellulaires, métaboliques, génétiques, …)
- Suivi de cellules / agent pathogène, interactions
- Analyses structurales et/ou fonctionnelles
- Biodistribution de molécules
- Evaluation de l’efficacité de candidats thérapeutiques
- Etudes pharmacodynamiques, pharmacocinétiques
- …
- Suivi de processus physiopathologiques (croissance tumorale, inflammation,
Localisation : Campus Institut Pasteur Lille - Animalerie IPL
Contact : Sophie Salomé-Desnoulez | 03.20.87.10.40